摘要
生物成像是我们了解从原子到有机体的生命系统的主要工具之一。成像技术的快速发展提高了我们检查这些系统的空间和时间分辨率,也使更大组织体积的可视化成为可能。这些进步具有巨大的潜力,但也产生了越来越多的必须存储和分析的成像数据。公共图像存储库通过开放数据提供提供关键的科学服务,支持科学结果的可重复性,访问参考成像数据集,为新的科学发现和加速图像分析方法的开发重用数据。图像数据的规模和范围为图像数据的开放共享提供了挑战和机遇。在这篇文章中,我们提供了一个受几十年来提供的生物信息开放数据资源影响的视角,提出了需要关注的领域和实现全球互操作性的途径。
介绍
生物成像作为理解生物系统的关键方法有着悠久的历史。从早期的手工磨片显微镜到现代高度自动化的显微镜,成像技术与我们对生命本身的理解携手并进。自计算显微镜出现以来,改进的传感器技术,自动化和缩放使得从许多不同模式收集的图像数据量不断增加。
不仅数据量迅速增加,而且从成像数据中获得科学结果越来越需要复杂的分析管道,这些管道作用于原始图像以最终产生信息(Bagheri et al. 2022)。这些管道本身也在快速发展,人工智能(AI)方法从根本上改变了生物图像分析(von Chamier et al. 2019)。
除了数据量的增长之外,社区要求公开成像数据的压力也越来越大,这既是为了支持再现性,也是为了利用现有数据进行新的研究。
开放数据共享的好处
图像数据的开放共享有几个好处,特别是当数据根据可查找、可访问、可互操作和可重用(FAIR)原则共享时(Wilkinson et al. 2016)。这些好处包括:
-
它通过提供产生定量结果的原始和衍生图像的访问,特别是当图像数据与用于得出这些结果的分析方法相关联时,支持科学手稿基础分析的可重复性。
-
随着时间的推移,它允许构建整理数据的集合,为特定领域的成像或生物学研究提供社区标准参考。例子包括细胞或组织图谱。
-
它减少了重复成像实验的需要,节省了时间,降低了成本,减少了在研究中使用动物的需要。
-
它通过为新方法的培训、验证和测试提供数据,加速了图像分析方法的开发和改进。
除了对科学界的好处之外,开放数据共享也给研究人员个人带来了好处。这些措施包括提高工作的可见性,以及不同于主要手稿出版的引用和重用途径(Piwowar和Vision 2013)。
EMPIAR和生物图像档案:背景
欧洲分子生物学实验室的欧洲生物信息学研究所(EMBL-EBI)为生物研究的许多领域提供公共数据资源。这包括生物成像,电子显微镜公共图像档案(EMPIAR, https://www.ebi.ac.uk/empiar/)和生物图像档案(BIA, https://www.ebi.ac.uk/bioimage-archive/)一起提供任何形式、任何规模的成像数据的免费公共档案。
EMPIAR (Iudin et al. 2023)成立于2013年,最初用于保存用于结构生物学的电子冷冻显微镜(cryo-EM)体积下的原始图像,地图和层析图本身存储在电子显微镜数据库(EMDB;Lawson等人,2016)和蛋白质数据库(PDB;wwPDB联盟2019)。在社区需求的推动下,EMPIAR的范围已经扩大到包括体积电子显微镜(vEM) (Peddie et al. 2022),以及软硬x射线断层扫描。
BioImage Archive (Hartley et al. 2022)于2019年启动,此前社区呼吁为生物成像数据提供通用存储库(Ellenberg et al. 2018)。BIA接受来自任何没有更专业资源的成像模式的数据(例如EMPIAR的3D电子显微镜数据),并建立在通才生物研究资源(Sarkans等人,2018)。它作为沉积数据库,旨在支持增值资源,如图像数据资源(Williams et al. 2017),该资源提供高度整理的参考成像数据集。
数据增长和多样性
EMPIAR和BIA自最初推出以来发展迅速(图1)。在撰写本文时(2023年5月),这两个资源加起来使科学界可以公开访问近3pb的成像数据。
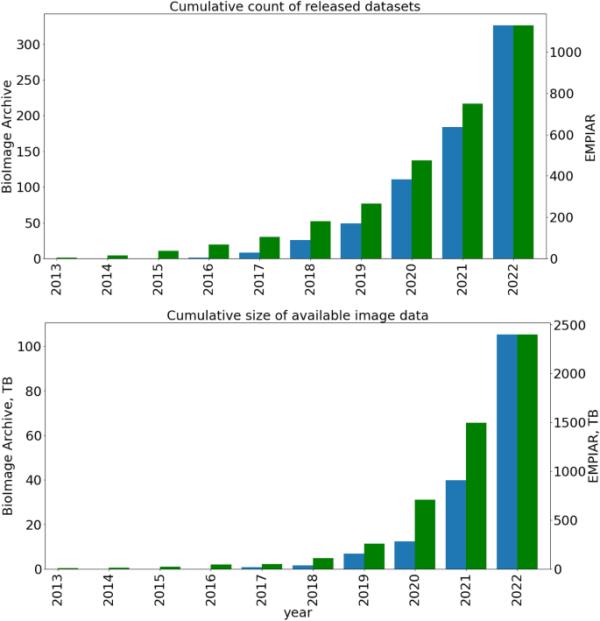
EMPIAR(绿色)和BioImage Archive(蓝色)发布的数据集总数(上表)和可用图像数据的累积大小(下表)的增长
除了规模之外,档案馆藏品中所代表的成像模式的范围也在迅速增长。BioImage Archive的通才角色是目前可重复使用的20多种不同成像模式的成像数据。虽然EMPIAR最初的重点是低温电镜,但增加了许多组成低温电镜的技术,大大增加了成像类型的多样性。
框1:EMBL-EBI数据资源
EMBL-EBI的数据资源支持广泛的生物领域;参见下面的表1中的示例。这些资源跨越了(a)数据量非常大的资源[包括那些拥有100s pb数据的资源,如欧洲核苷酸档案(ENA)和欧洲基因组-表型档案(EGA)];(b)长期存在的[如欧洲蛋白质库(PDBe),它是世界蛋白质数据库(wwPDB)的一部分,该资源成立于1971年,现在已有50多年的历史];(c)在其领域中是独一无二的[例如全基因组关联研究(GWAS)目录]。
这些资源每月从超过500万个唯一的互联网协议(IP)地址接收超过30亿个web请求。它们在全球范围内使用,2021年联合国(UN)每个成员国都有访问记录。许多资源是国际联盟的主要成员,如国际核苷酸测序联盟,wwPDB或ProteomeXchange联盟。它们共同为全世界的科学家提供了巨大的价值。
目录
摘要 介绍 大规模公平:挑战 从挑战到机遇 展望未来 有限公司 包括备注:承诺开放、公平成像数据 数据可用性 参考文献 致谢 作者信息 道德声明 相关的内容 搜索 导航 #####大规模公平:挑战
在这种规模上提供对成像数据的访问暴露了许多挑战,其中一些是成像领域独有的,而另一些则更为普遍。
生物成像是异质的
生物成像是一个宽泛的术语,涵盖了非常广泛的成像技术,包括基于光的显微镜以及电子显微镜、扫描探针显微镜等的许多变体(图2)。这些技术中的每一种本质上都是一个自己的领域,有自己的社区、标准、实践和方法。它们也发展迅速,因此需要新的分析工具和方法,以及新的术语和元数据标准来理解和组织生成的图像。
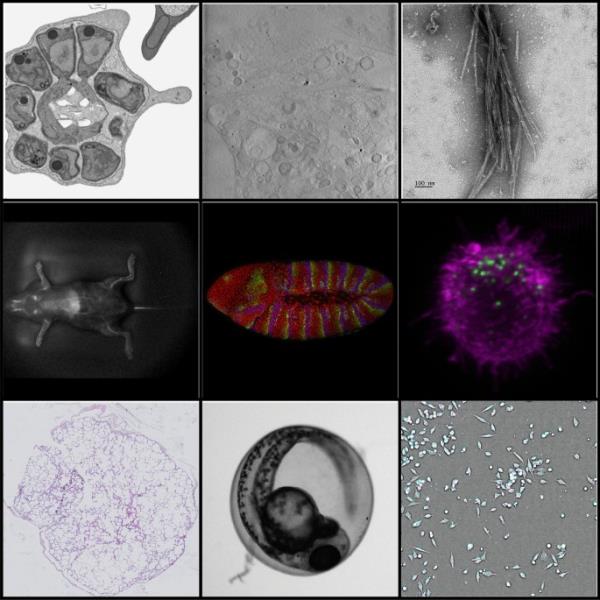
成像应用/模式的多样性。从左到右,从上到下:聚焦离子束扫描电子显微镜(FIB-SEM);EMPIAR-10310)、软x射线断层扫描(EMPIAR-10416)、二维电子显微镜(S-BIAD462)、微波红外成像(S-BIAD548)、共聚焦荧光显微镜(S-BIAD582)、点阵光片显微镜(S-BIAD573)、组织学全切片成像(S-BIAD419)、宽视场显微镜(S-BIAD531)和多通道高含量筛选(S-BIAD145)
这些捕获方法的差异,以及它们在不同仪器中的实现,产生了许多不同的文件格式,图像数据可以用这些格式表示。对于那些希望查看、访问和重用开放成像数据的人来说,这种文件格式的混乱是一个重大障碍。
此外,公共图像数据资源必须组织这些不同类型的图像,以确保它们仍然是可查找的。组织数据需要高质量的元数据(linkert et al. 2010)。然而,生物成像技术的这些异质性使得建立一致的元数据标准变得更加困难。
在几个轴上成像数据很大
正如我们所展示的,EMPIAR和BioImage Archive提供的数据量正在迅速增长。这反映了所管理的图像数据量的普遍增长,这带来了不同的挑战。
数据总量
随着个人资料的增长,大量的数据必须从提交者转移到存档(或在机构之间进行数据的出版前管理)。EMPIAR最大的单个数据集的大小超过70tb,这可能需要数周的时间来传输,特别是因为网络带宽不能按照数据增长的速度进行扩展。在数据文件上运行的自动检查(例如,确定图像的完整性)通常必须读取整个图像,这与图像大小成线性比例。扩展存储以应对如此大的数据量也需要大量的财务和基础设施投入,尽管有规模经济可以帮助大型机构数据提供商。
非常大的个体图像也具有挑战性。如此大的个体图像来自不同的捕获方法——vEM技术可以产生非常大的图像,2D电子显微镜图像拼接技术也可以产生非常大的图像(Faas et al. 2012)。薄片显微镜,特别是跨多个荧光通道的长时间序列,也产生非常大的单个图像。这些单个图像的大小可以达到数百gb到tb。它们很难在计算机内存中使用,并且在共享时,如果用户需要下载整个映像才能使用它,则很难访问它们。
大型成像数据集有时被结构化成非常大量的单独文件,要么是因为这些数据集涉及由高度自动化过程(例如高通量显微镜)捕获的多个图像,要么是因为每个切片图像单独存储的切片技术。这些是传统计算机文件系统难以处理的;例如,包含数百万个单独文件的目录会导致严重的性能问题。这样的数据集还需要仔细选择存储元数据的方式,因为支持跨数百万个文件的搜索可能具有挑战性。
最后,必须管理的单个数据集的数量是一个挑战。通常需要人工管理数据集,以确保遵循适当的标准。这种管理所需的时间与必须审查的数据集的数量成线性关系。此外,随着数据集变得越来越复杂,整理所需的时间也在增加,例如对于下面描述的相关或多模态数据,整理需要多个不同生物数据领域的专业知识。
图像数据通常与其他数据类型集成在一起
生物成像增加的复杂性部分来自于多模态。跨尺度成像通常需要相关的方法,例如,结合荧光显微镜来识别感兴趣的区域,然后使用电子显微镜进行详细的探索(Sartori et al. 2007)。这允许覆盖大样本量的组合,同时在需要的地方提供高分辨率。
成像数据越来越多地与其他类型的生物学数据相结合,如转录组学、蛋白质组学或代谢组学数据。这些“空间组学”技术允许对关键生物过程进行精确的物理和时间定位。然而,这种数据集成使得一致的数据标准变得更加重要,因为集成依赖于识别和分类。对于筛选数据集,链接到ChEMBL等知识库(Gaulton et al. 2017;Mendez et al. 2019)和UniProt (UniProt Consortium 2023)对于筛选的化合物或蛋白质鉴定的一致性通常具有额外的重要性。
归档和提供对这些数据集的访问也是一个挑战。通常,档案专门针对不同的数据类型——例如,EMBL-EBI分别提供基因表达数据(Moreno等人,2022)、蛋白质组学(Perez-Riverol等人,2022)和代谢组学(Haug等人,2020)的档案。对于多模态数据,必须考虑是否以及如何分割或复制传入数据,以及作为一个统一的整体呈现(图3)。
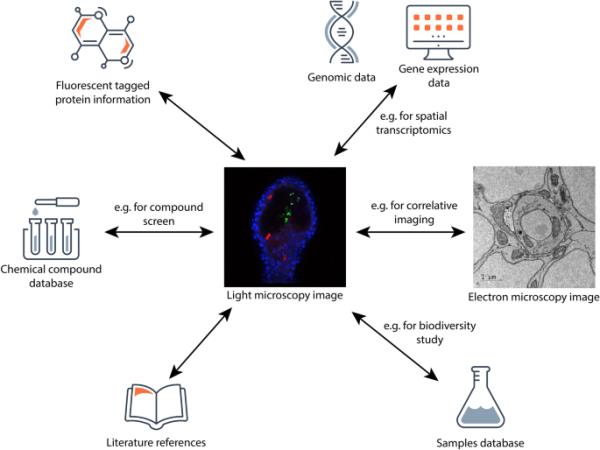
以一种对再现和重用有用的方式描述图像通常涉及到对其他类型生物信息的引用和链接。中央图像来自S-BSST429,“斑马鱼作为刚地弓形虫复制和与巨噬细胞相互作用的体内研究的新模型”(Yoshida et al. 2020),右侧是EMPIAR-10460的相应图像。这些其他生物领域的专门数据资源可以提供专用功能,例如,用于样本管理的BioSamples (Courtot等人,2022),用于基因组序列数据的欧洲核苷酸档案(Cummins等人,2022)和用于荧光蛋白的荧光蛋白数据库(FPbase) (Lambert 2019)。
从挑战到机遇
这些挑战也带来了机遇——与其他生物数据类型紧密集成的更全面、更高分辨率的成像数据具有巨大的潜力,可以促进我们对生物系统的理解(Lewis et al. 2021)。通过关注格式和元数据的一致数据标准,以及社区的密切参与,管理异构性和规模成为可能。
标准化的数据格式和使用适当的存储
通过适当使用现代云优化的文件格式,可以减轻大型图像的挑战,特别是使它们易于浏览和访问,这些文件格式旨在适应对图像组件的快速访问,并包括预先计算的下采样表示,以允许快速预览。OME-NGFF文件格式(Moore et al. 2021)提供了所有这些功能,并为广泛采用提供了强有力的支持(Moore et al. 2023)。使用对象存储以这些数据格式托管映像,既提供了一种扩展到非常大的存储系统的方法,也提供了一种数据访问协议,允许更容易地共享结构化数据。
选择数据存储技术需要在成本、容量、速度、冗余/弹性、访问协议和其他因素之间进行大量权衡。对象存储单位容量通常比文件系统存储便宜(例如,截至2023年5月,Amazon Elastic File storage比同等S3对象存储贵大约10倍)。然而,数据访问模式各不相同,一些数据集的访问频率比其他数据集高得多;因此,可以根据访问模式对存储解决方案进行调优。特别是,我们可以有效地将更快的存储用于最近和更频繁访问的数据集,而将更便宜、更大容量的存储用于其他数据集。对于很少访问的数据,图像可以保存在“冷库”上,比如磁带。这种“分层”存储方法为实现长期可持续性铺平了道路。
通往和谐之路nis我tadata
早期关于图像元数据的关键工作为图像文件开发了标准和模型(Allan et al. 2012),通常侧重于与图像采集过程相关的元数据以及图像数据如何在文件中结构化。这项工作的后续扩展对特定领域(如光学显微镜)的模型进行了改进和改进(Hammer et al. 2021)。
要了解成像实验结果的背景,还需要了解有关实验目的、成像样本及其制备、如何分析图像以及如何与其他数据源联系的信息。生物图像推荐元数据(REMBI);http://k1.fpubli.cc/file/upload/202308/21/ghcjvotmf55。是一套由更广泛的生物成像社区内不同亚组的代表制定的指南(Sarkans等人,2021)。它为成像元数据提供了组织原则,这些元数据捕获了实现图像重用所需的所有上下文(图4)。
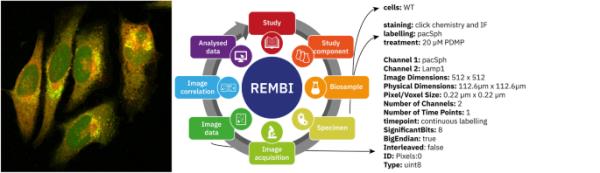
显示图像以及与REMBI模型对齐的元数据,包括图像文件结构、样品处理和荧光通道内容,来自BioImage Archive登录S-BIAD144 (Hartwig和Höglinger 2021)。
总之,这些详细的模型和组织原则为协调不同图像数据存储库中的元数据模型提供了一条途径。对模型组件的共享理解支持元数据的映射和更好的互操作性。这对于集成多模态数据尤其重要,因为跨数据域使用标准化和共享的标识符至关重要。
与社区合作
就像成像本身一样,生物成像社区是庞大而多样的。不同的生物成像领域往往有自己的亚群体,有独立的网络、会议和首选的传播途径。然而,跨越国家、大陆和国际领域的成像组织,如欧洲生物成像、北美生物成像和全球生物成像,为这些社区聚集在一起提供了焦点。
成像科学家在这个群体中扮演着关键角色。他们直接与实验生物学家合作,进行假设驱动的研究,并支持和开发成像仪器技术。他们的参与对大规模图像数据共享的长期成功至关重要。应该通过提供指导、工具、培训和文档来支持成像科学家,以帮助注释和共享精心策划的成像数据集。在这些科学家致力于支持和培训其他人的地方(例如,在成像设施中),“培训培训师”的方法能够扩大良好实践的范围。此外,他们的反馈和指导对于那些试图开发用户友好的系统来提交、管理和重新使用图像数据的人是至关重要的。
生物图像分析社区是图像数据难题的关键部分(Cimini et al. 2020)。方法开发人员通过发布他们的工作成果来贡献数据资源,并从对数据的开放访问中获益,从而测试和验证他们的方法。提供培训课程的机构经常利用公共数据进行演示。序列和结构数据资源随着生物信息学社区不断变化的需求而得到支持和发展;同样,成像数据资源和生物图像分析人员可以相互提供很多东西。
最后,随着更广泛的用于管理图像数据的共享资源上线,有大量的机会可以有效地协同工作。大型档案馆可以与地方和国家机构合作,就标准达成一致,开发出版渠道,并在工具和方法上进行合作。随着越来越多的国内和国际数据资源的出现,有可能将它们连接在一起,以共享大规模数据提供的需求。
从仪器到档案:出版前的图像数据
出版前的数据管理,从项目规划到捕获和初步分析,与公共数据共享的目标有些不同。然而,这两个领域在挑战和方法上都有相当大的重叠。特别是,格式的多样性、元数据标准化的差距和数据量的庞大规模都造成了困难。在必须跨协作组或团队共享数据的情况下,数据传输的挑战也同样适用。
幸运的是,支持良好的开源社区工具可以解决其中一些挑战,为希望管理其数据的机构和设施提供了一个起点。这些工具包括OME远程对象(OMERO);Allan et al. 2012), XNAT (Herrick et al. 2016)和Cytomine (marsamade et al. 2016)以及许多其他平台,提供存储,组织和可视化图像的平台,以及管理用户帐户和提供不同程度的集成分析软件。
除了这些现成的解决方案之外,图像数据格式和元数据模型的标准化还为发布前数据管理和公共存储库之间的共享构建块提供了机会。在这个共享的基础上,可以构建管理数据移动的工具,与分析软件和数据注释/管理集成。
敏感的哒答:更多的挑战和更多的好处
本文的主要重点是共享不敏感的图像数据(即不包括患者可识别信息的图像数据)。共享敏感数据的挑战使上述困难与管理受控访问基础设施的法律、道德和监管要求相结合。然而,能够共享敏感的人体成像数据显然有巨大的潜在好处,包括开发新的临床方法和诊断工具。
欧洲基因组-表型档案(EGA;Freeberg et al. 2022)提供永久存档和共享个人可识别的遗传、表型和临床数据的服务。虽然目前EGA的大多数用途是共享遗传数据,但有可能与以成像为重点的数据资源共同开发,以支持与遗传数据相关的成像数据的存档和分发,或者将EGA开发作为以图像为重点的敏感数据资源开发的模式。这项工作最初可能通过结构化数据生成项目获得资金,然后扩大规模。
展望未来
生物学中其他数据领域的经验教训
EMBL-EBI提供跨生物数据许多领域的数据资源(Thakur et al. 2023)。其中包括蛋白质数据库(wwPDB Consortium 2019)等数据库,这些数据库已经存在了50多年,实际上与计算生物学本身一样古老。大规模管理生物数据资源的经验给我们带来了一些教训,这些教训与未来管理全球成像数据的路径有关。
即时通讯准则和标准的重要性
使用元数据建议,如微阵列实验最小信息(MIAME) (Brazma et al. 2001)或测序实验最小信息(MINSEQE) (Brazma et al. 2012),以及实验因子本体(Malone et al. 2010)等本体,提高了数据的互操作性,特别是允许跨实验进行比较。这对于大规模再分析和有效共享至关重要。中央档案资源作为这些标准的守护者和倡导者发挥着重要作用,没有这些标准,就很难始终如一地执行。REMBI指南为填补生物成像的这一角色提供了一个起点。
简单
在支持数据交换和发布方面,科学界和项目的资源有限。最初复杂的标准实现(Spellman et al. 2002)有陡峭的学习曲线,但随着时间的推移会从简化中受益(Rayner et al. 2006)。更简单的标准提供了更容易和更广泛的采用,这反过来又支持了这些标准的进一步开发。
分离数据归档和增值资源
建立一个成功的科学数据档案的主要原则是:捕获实验数据和支持元数据,即科学记录;提供简单的数据提交流程;协助制定标准,并采用现有标准;不要进行数据价值判断或复杂的数据整合。增值资源可以丰富、组合和管理数据集;再加工数据;运行分析;等。这两组功能非常不同,需要不同的资源治理、流程和技术基础设施方法。
需要与国际社会共同成长和适应
随着数据资源的发展,对大规模协作来分担管理负担的需求也在增长。国际核苷酸序列数据库协作(Cochrane et al. 2011)和全球蛋白质数据库(Berman et al. 2003)是随着各自生物领域的成熟和对联合提供资源的需求的增长而产生的。对于数据消费,与数据分析师合作并了解需要支持哪些有用的数据访问模式非常重要。
数据集成价值
个别实验的结果不会存在于真空中。通过成像研究的系统、生物体、过程、基因、化合物、组织和细胞通过多种研究途径进行检查。如上所述,当与其他类型的数据相结合时,成像数据通常是最有信息量的。确保以允许链接到这些其他数据的方式记录图像元数据支持此类数据集成。一个例子是在本地化研究中使用一致的蛋白质标识符,这样它们就可以链接到蛋白质序列数据库,如UniProt (the UniProt Consortium 2023),并从那里链接到许多其他资源。
数据重用中的长期主义
除了立即重用案例之外,提供组织良好的生物数据可以带来意想不到的好处。基于AlphaFold (Jumper et al. 2021)的AlphaFold蛋白质结构数据库(Varadi et al. 2021)彻底改变了结构生物学。自1971年PDB成立以来,AlphaFold建立在多年的精心管理、标准开发和社区对共享存档的鼓励之上,以及电子数据库之前更老的UniProt前身(Dayhoff 1969)。在创立之初,AlphaFold成功背后的深度学习方法还不存在,很少有人能预测到半个世纪后它们会变得如此成功和重要。
提供大规模的公平图像数据:前进的道路
考虑到云准备成像格式、元数据模型的发展以及其他生物数据领域的指导所带来的可能性,我们可以提出下一步的建议,以支持图像数据和元数据的大规模开放共享。
致力于为发布提供一组标准图像格式
随着社区聚集在一组可以满足数据生成器、分析人员和存款人需求的图像格式上,围绕它们建立一个丰富的工具和资源生态系统成为可能。除了减少对数据转换的需求外,这些格式还为在线数据访问提供了标准化协议,减少了耗时的下载需求,并实现了快速可视化。
就处理图像数据的共享词汇表达成一致
受控词汇表和完整本体是支持数据集成和比较的关键组件。关于在管理成像数据的社区中使用哪一种协议将是实现互操作性的良好的第一步。
发展和成长共享核心我tadata标准
尽管不同数据管理系统的需求不同,但基于REMBI原则的共享元数据核心集达成一致将为互操作性提供基础。一个最初的核心集可以在现有的数据提供者之间达成一致,并随着时间的推移而扩展。
跨规模调整数据管理系统
一旦统一了标准化数据格式、共享词汇表和元数据模式,管理成像数据的数据资源之间的协作就变得容易得多。数据可以在发布前数据管理系统和公共存储库之间流动,并且可以跨大型档案进行联合。
下载原文档:https://link.springer.com/content/pdf/10.1007/s00418-023-02216-2.pdf为您推荐:
- 慈善机构梦想在一个创造性的筹款努力 2024-05-11
- 以前的Twitter X可以让用户隐藏曾经引以为豪的蓝勾 2024-05-11
- 挪威公主放弃皇室职责,专注于与未婚夫的生意 2024-05-11
- 我的政府将解决电力部门的赤字——蒂努布 2024-05-11
- 北约领导人担心乌克兰冲突可能会蔓延 2024-05-11
- 德桑蒂斯在推特上与埃隆·马斯克对话,宣布将参加2024年总统竞选 2024-05-11


